6️⃣分钟获得拟南芥的全基因组结果!

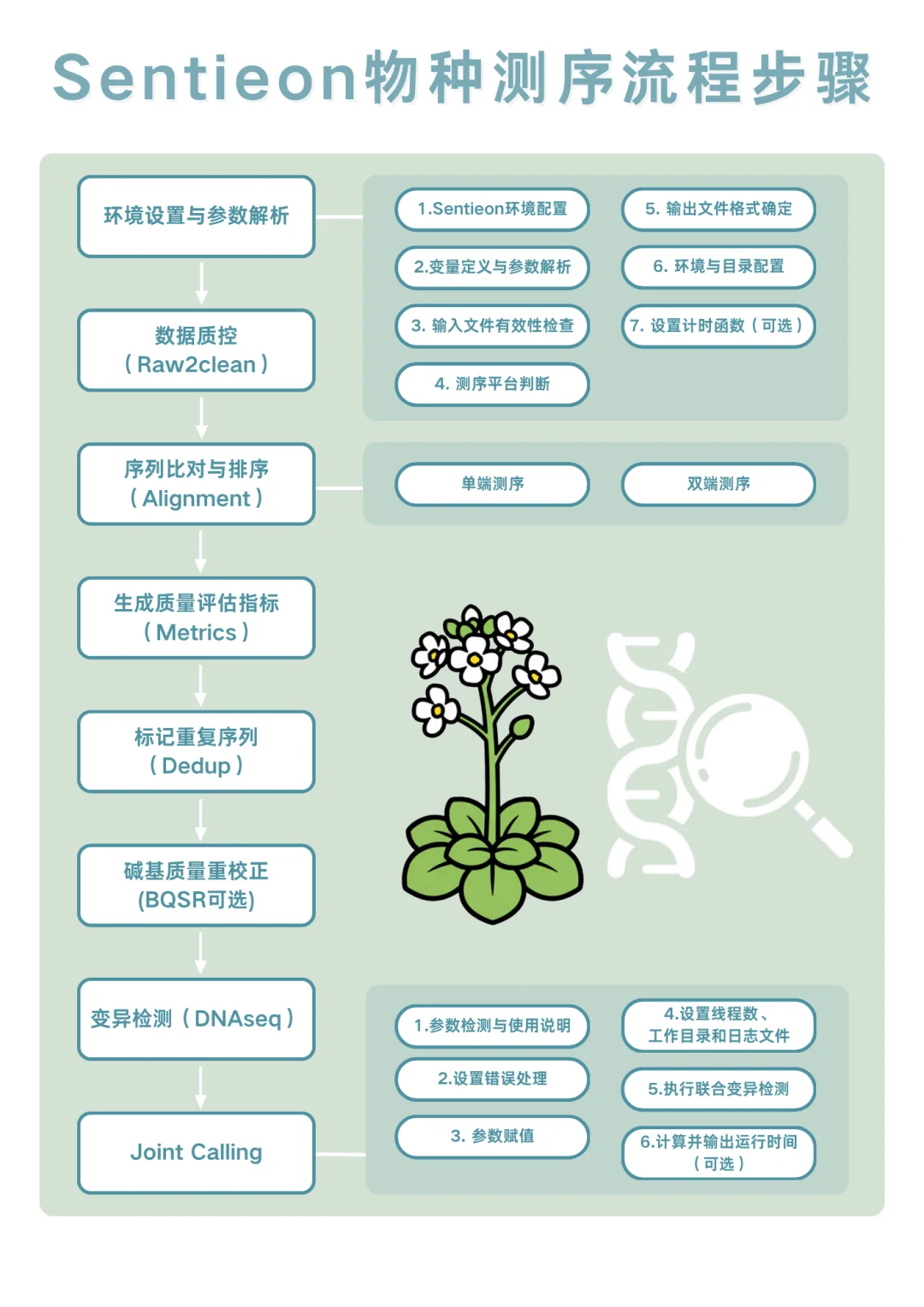



💻📊基于 Sentieon 软件开发的用于拟南芥的全基因组测序数据的自动化流程脚本。 该流程实现了从原始测序数据(FASTQ)到变异检测结果(GVCF)以及joint calling的完整分析流程,支持多个测序平台和输出格式。
📝脚本支持原始测序数据(raw_fastq)、过滤后的测序数据(clean_fastq),进行质控、比对、排序、标记重复、生成质量评估指标,通过Haplotyper 算法进行变异检测,输出 gVCF 文件。
自动化:自动检测测序平台和参考基因组以决定合适的分析策略。
稳健性:使用 set -euxo pipefail和标记文件实现错误处理和断点续跑。
模块化:将每个分析步骤封装成函数。
高效性:使用商业优化的sentieon工具替代金标准GATK/BWA,速度更快。
可追溯性:详细的日志记录和 MD5 校验确保结果可重现。
⚠️要运行此脚本,需要预先安装好 sentieon、fastp等软件,并准备好对应的模型文件 bundle。
📝脚本支持原始测序数据(raw_fastq)、过滤后的测序数据(clean_fastq),进行质控、比对、排序、标记重复、生成质量评估指标,通过Haplotyper 算法进行变异检测,输出 gVCF 文件。
🧬🧬该脚本适用于大规模基因组变异检测场景。 特点包括:
自动化:自动检测测序平台和参考基因组以决定合适的分析策略。
稳健性:使用 set -euxo pipefail和标记文件实现错误处理和断点续跑。
模块化:将每个分析步骤封装成函数。
高效性:使用商业优化的sentieon工具替代金标准GATK/BWA,速度更快。
可追溯性:详细的日志记录和 MD5 校验确保结果可重现。
⚠️要运行此脚本,需要预先安装好 sentieon、fastp等软件,并准备好对应的模型文件 bundle。
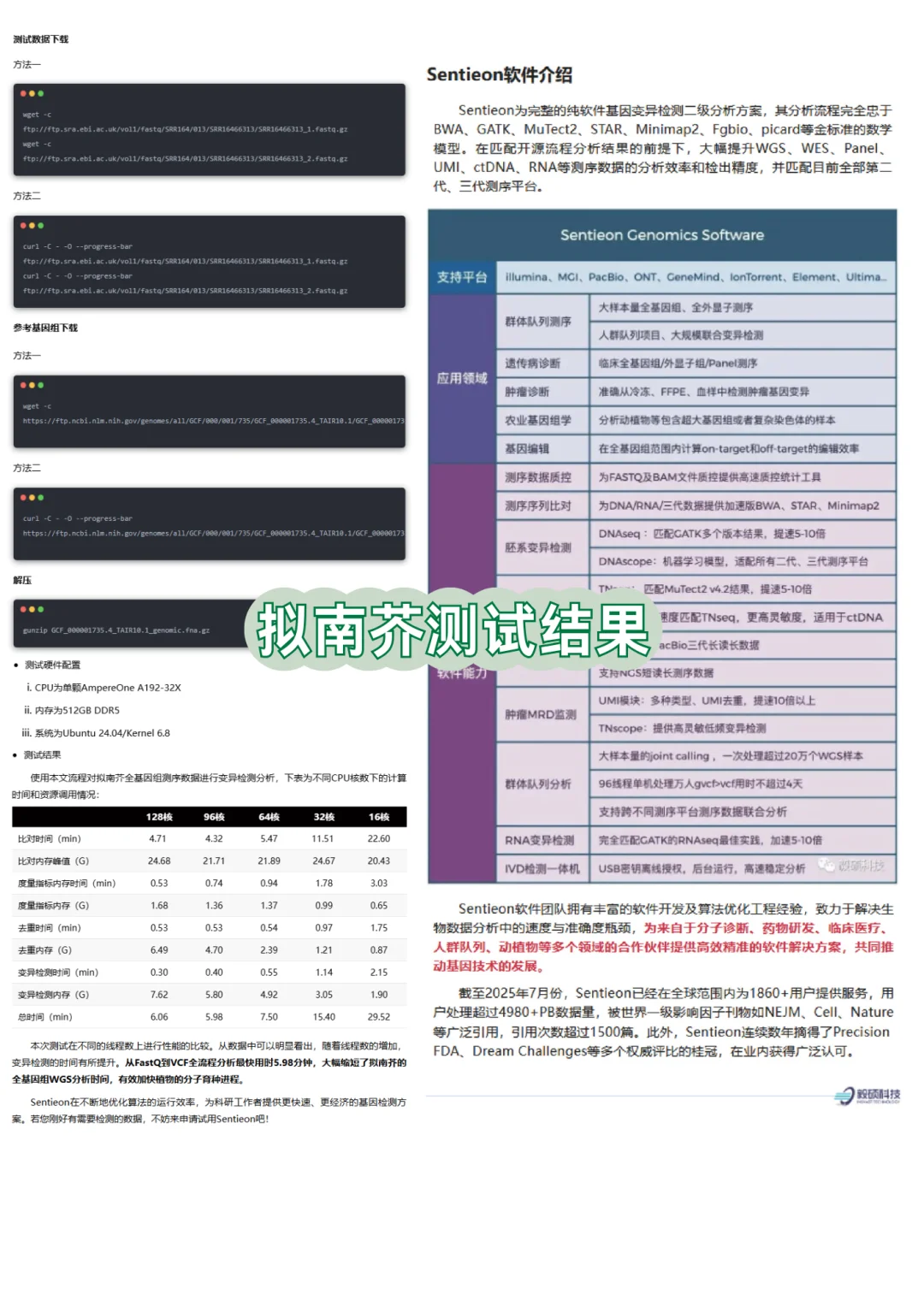
🌱本次测试在不同的线程数上进行性能的比较。从数据中可以明显看出,随着线程数的增加,变异检测的整体效率显著提升。
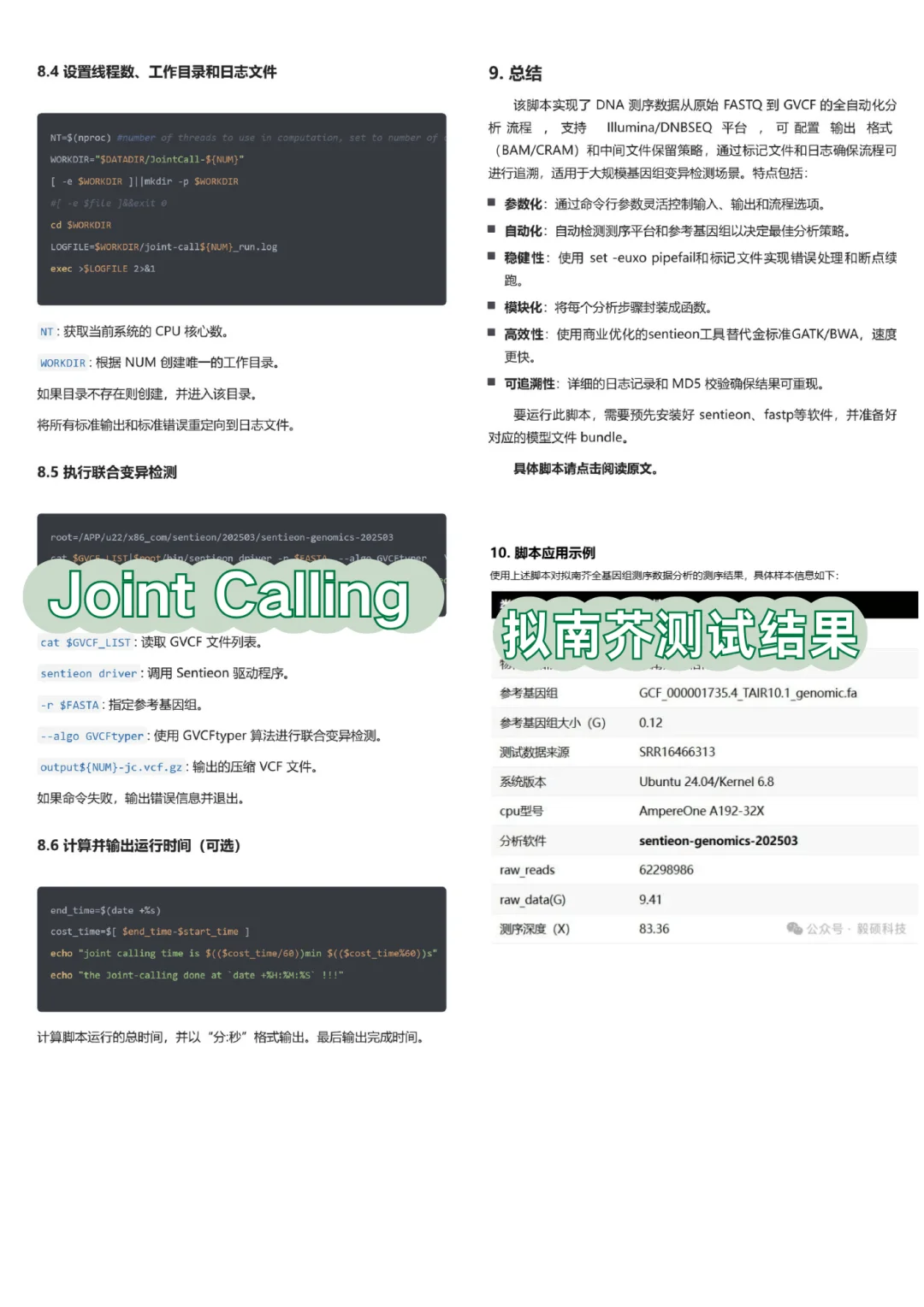
⚡⚡从FastQ到VCF全流程分析仅用时5.98分钟,大幅缩短拟南芥的全基因组WGS分析时间,有效加快植物的分子育种进程。
🚀Sentieon在不断地优化算法运行效率,致力于为科研工作者提供更快速、更经济的基因检测方案。若您刚好有需要检测的数据,不妨来申请无偿试用Sentieon吧!
💪 👏 基因智算工坊:不做测序,不做科服,专注Call SNP/Indel/SV,日均3000样。
 夜雨聆风
夜雨聆风





