插件 | 应用 GPT 开发「计算蛋白质理化性质参数」工具
写在前面
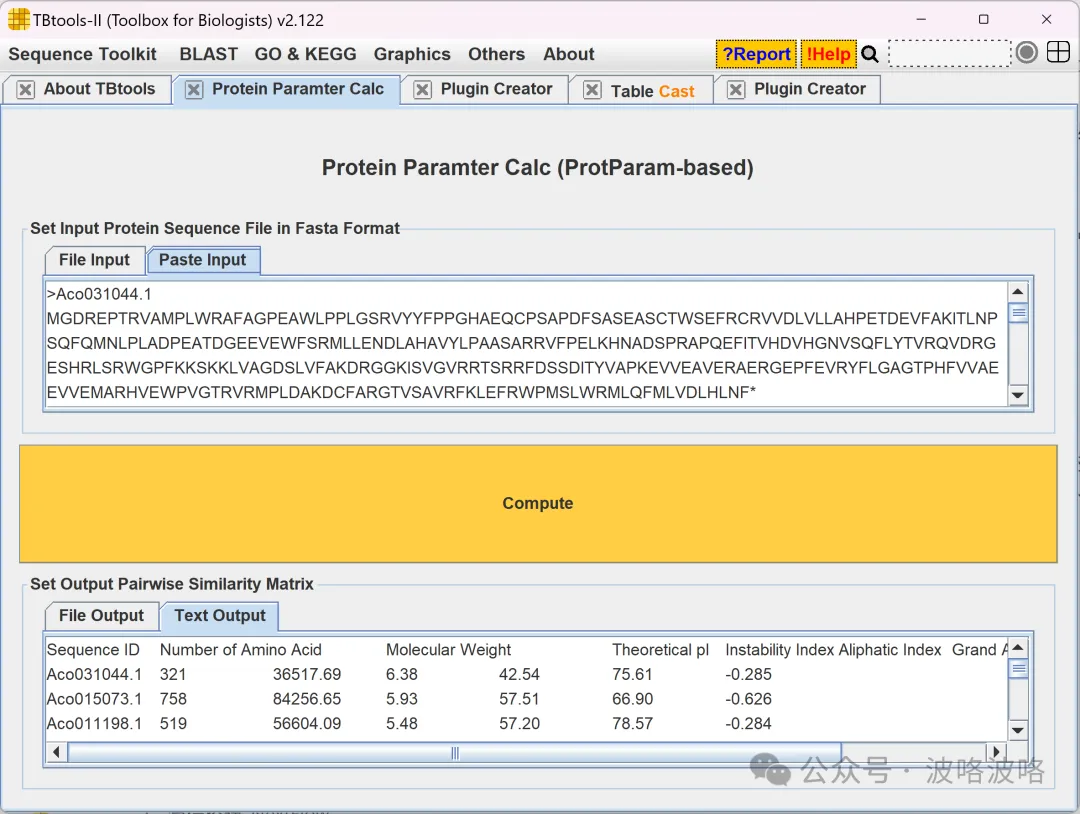
早前,我在 TBtools 中实现了一个基于网页爬虫的功能,通过逐个序列提交到 「Expasy ProtParam」来实现批量蛋白质序列参数,如分子量,等电点,芳香系数,不稳定系数,等等。
但是这个功能存在明显问题,也就是基于网络。网络可能不稳定,尤其是一些地域直接无法访问该服务网站。
为此,我后续有应用 GPT 开发了一个 Python 插件,这个插件基于 BioPython,所以出来体积比较大,大概 132 Mb。今天,我突发奇想,现在 o1-previw 推理能力不错,应该能够更好的实现的计算方法。不如让他从零用 Java 写一个,这样插件大小就超级mini。
开发过程
大体我用了半个小时,一边和朋友聊天,一边和GPT聊天最终解决问题。主要开发过程简述如下:
-
先用 o1-preview 模型直接写出 java 的分析脚本,对照正确结果,发现不对就让他优化,o1-preview 基本都能优化,不过可能出现优化了第一个指标,结果第二个指标反而计算错,所以需要优化提醒他两三次;
-
由于其中有一个不稳定系数需要使用一个 400个二肽氨基酸对的稳定性指标,所以怎么让 o1-preview 输出他都不愿意。而且数值可能也不对。于是让他提供了如何获取这些数据,他给了一个文献。
-
翻阅文献,找到是一个 1990 年的文献,只有PDF版本,且是图片模式的表格,为此,我用 GPT-4o 模型进行图片识别,但效果一般。最后还是让他建议一些转化png为表格的在线网站。使用这个网站,转换了表格,然后对照得到表格结果。
-
由于 o1-preview 需要的是代码中直接存储数据,而不是另外的表格,于是我直接给了数据,让他转换,很遗憾,还是因为数据太大,效果不佳。为此,我套用 o1-preview 模型让他生成java脚本,然后用 TBtools 插件开发模式直接转换表格。效果佳。
-
对应的数据置入原 o1-preview 开发额蛋白质参数计算脚本。效果良好。
最终插件开发界面如下
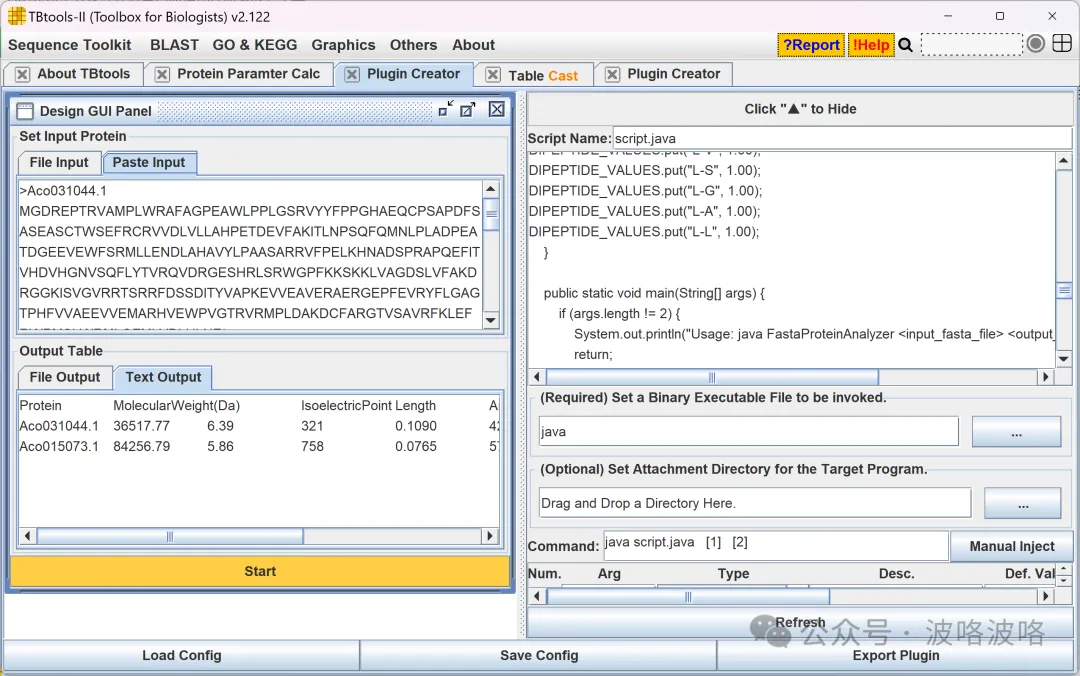
于是导出插件
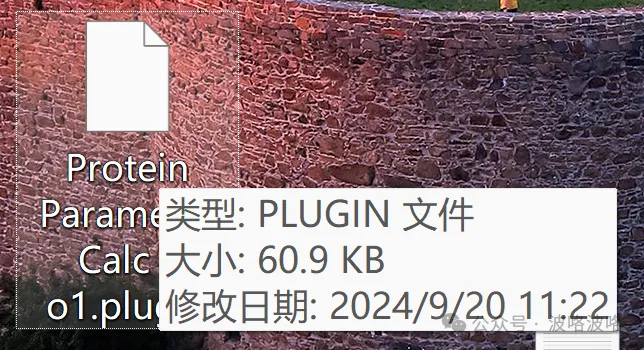
意不意外,惊不惊喜?只要 60.9Kb,相比于原来的 130Mb 的 python 插件,约等于无,但结果一样甚至更全面。
这是原始Java爬虫版本结果
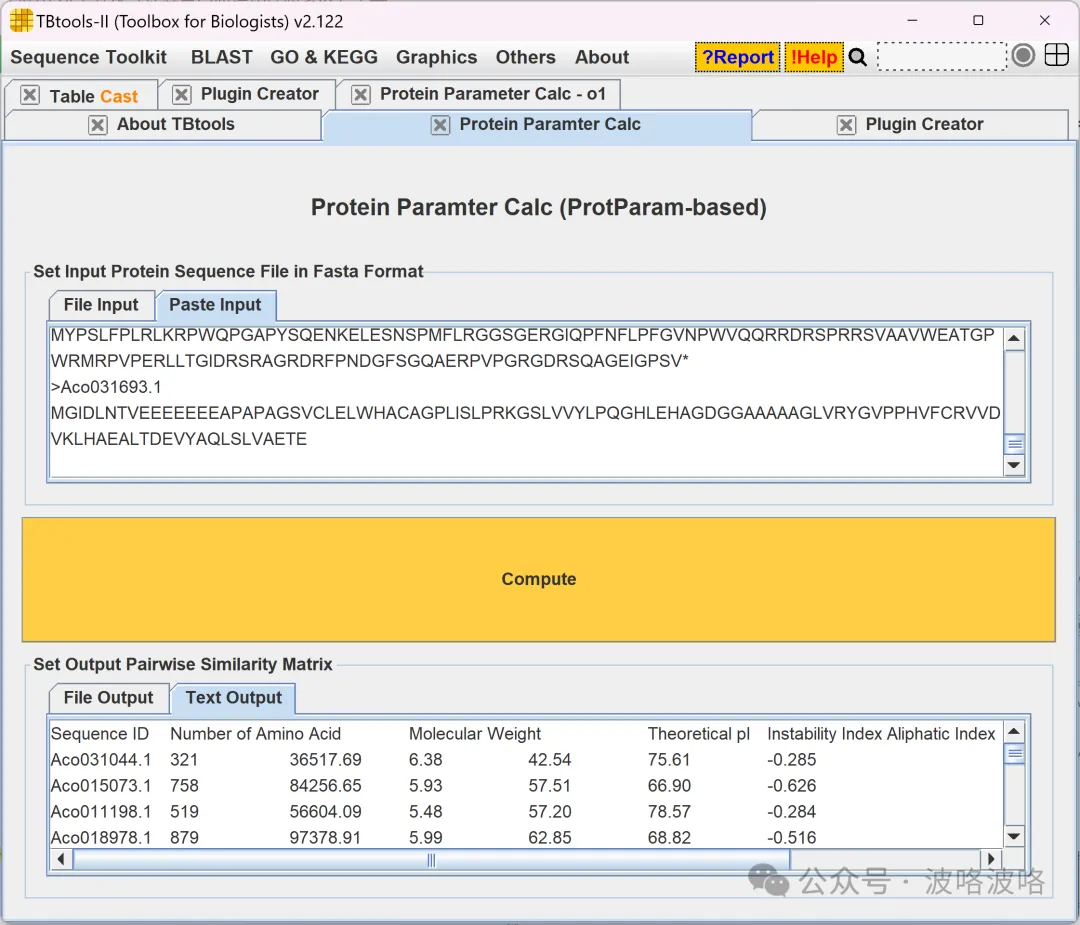
下述是 o1-preview 开发的版本计算结果。
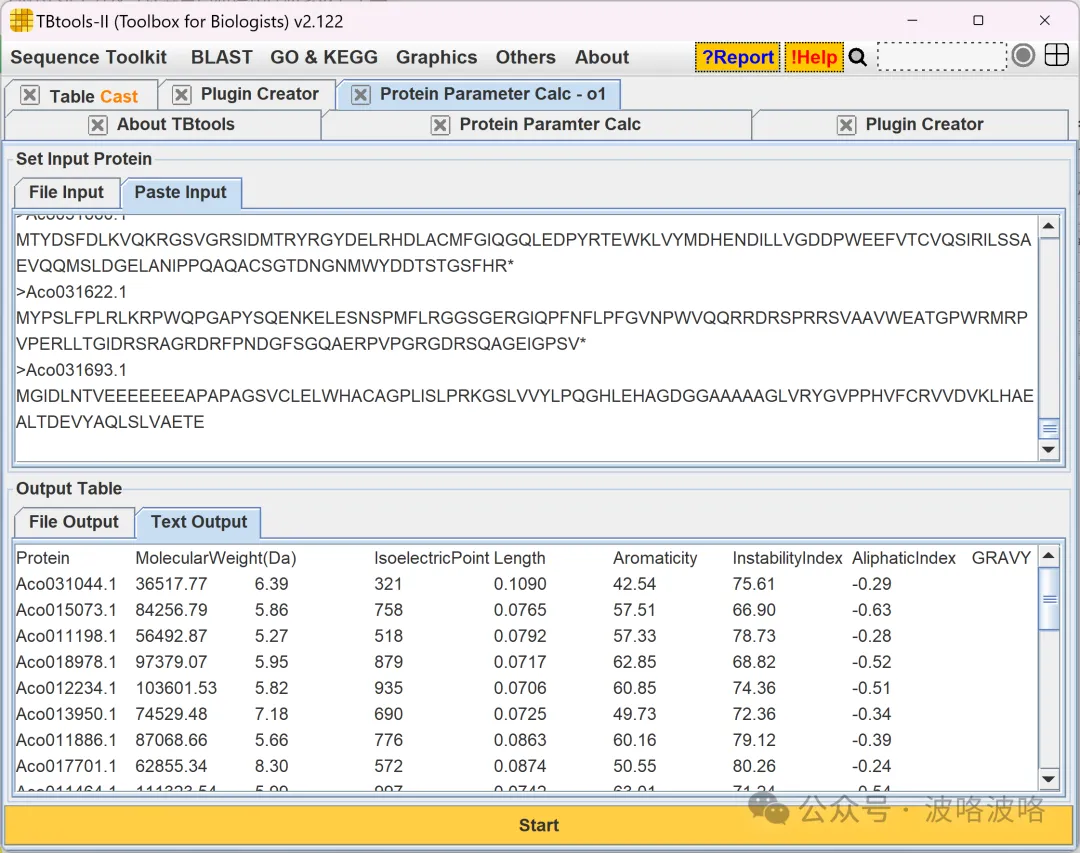
PS:Python 插件版本不纳入,实测不是很稳定,且体积过大。
写在最后
或许,其实有了 GPT,零基础就可以“造轮子”了。我们甚至不需要专门去学习一些计算公式和实现这些计算逻辑,直接让GPT生成可用代码,测试他的正确性和鲁棒性,然后以 TBtools 插件模式固定下来,从此以后我们就都可以使用了。
或许,对于很多人,只需要花自己10分钟的时间,就可以节省许多人的10分钟,四舍五入,约等于“救人一命”。
 夜雨聆风
夜雨聆风





